From PGI
(Created page with "= '''암 게놈에서 체세포 구조 변이의 다양성 메커니즘''' = 암 게놈에서 체세포 변이 식별이 NGS 분석을 통해 가속화되고...") |
|||
| Line 1: | Line 1: | ||
| − | |||
| − | + | = '''암 게놈에서 체세포 구조 변이의 다양성 메커니즘''' = | |
| − | |||
| − | |||
| − | | + | 암 게놈에서 체세포 변이 식별이 NGS 분석을 통해 가속화되고 있다. 그러나 구조 변이의 복잡한 특성을 알기에는 기본 메커니즘으로는 불충분하다. 생명의학 정보 센터와 하버드 의과대학 연구진들은 140명의 종양 환자들의 전장 게놈을 해독하여 체세포 변이를 예측하는 알고리즘에 적용하였다. 유전자 재배열과 같은 구조 변이들에 관여하는 돌연변이 메커니즘을 발견하였고, 이를 이용하면 염기서열 상에서 삽입과 결실, 복제 등의 차이를 규명할 수 있다. 특히, ''CDKN2A/B'' 유전자에서 염기 손실과 아교모세포종에 관련된 ''EGFR ''복제에 대한 메커니즘을 확인하였다. |
| − | |||
| − | |||
| − | + | 암은 단일 염기 변이(single nucleotide variation), 구조변이(structural variation), 염색체 이수성(aneuploidy) 등의 유전적 변화에 의해 발발한다. 특정 암 유형은 화학약물, 자외선, 흡연과 같은 것에 노출되어 특정 돌연변이와 복원 메커니즘이 고장 나서 발생하는 체세포 단일 염기 변이에 의해 분류된다. 큰 범위의 구조변이는 염색체 deletion, insertion, inversion, tandem duplication, translocation, complex rearrangement 등으로 인해 암에서 정상 유전자의 기능에 영향을 끼친다. 하지만 여전히 구조변이에 대해서는 밝혀진 것이 없다. | |
| − | |||
| − | |||
| − | + | === 체세포 구조변이를 식별하는 5가지 방법 === | |
| − | |||
| − | |||
| − | + | [[File:구조변이01.png|center|550px|구조변이01.png]] | |
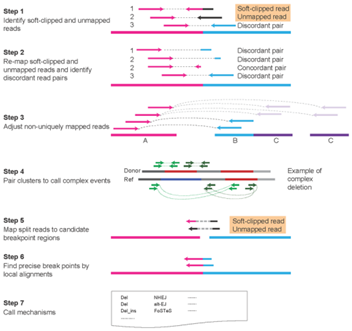
| − | | + | 첫 번째는, '''Meerkat을 이용하여 생식세포의 복잡한 결실을 규명'''하였다. 암에서 체세포 구조변이 돌연변이의 특징은 복잡하고 간단한 구조변이를 모두 규명할 수 있다는 것이다. '''Meerkat'''이라는 새로운 알고리즘을 이용하면, Soft-clipped과 매핑되지 않은 부분을 확인할 수 있고, discordant read도 확인할 수 있다. 따라서 특이하지 않은 부위의 매핑 read들을 이용하여 조정하거나 복잡한 것은 짝을 이뤄 클러스터 시킬 수 있다. 서열이 중단된 지역을 이용하여 염색체 중단이 일어난 지역을 유추하고, alignment하여 메커니즘을 불러 올 수 있는 것이다. |
| − | + | [[File:구조변이02.png|center|600px|구조변이02.png]] | |
| − | |||
| − | |||
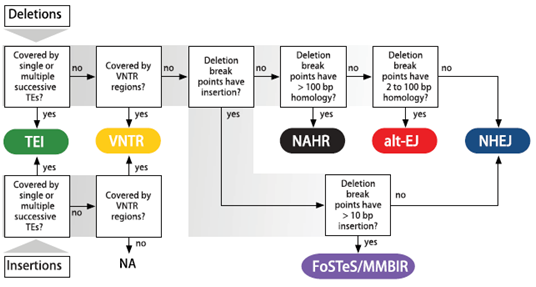
| − | | + | 이에 따라 6가지 분석 파이프라인으로 분류할 수 있는데, transposable element insertion (TEI), variable number of tandem repeats (VNTR), nonhomologous end joining (NHEJ), alternative end joining (alt-EJ), nonallelic homologous recombination (NAHR), fork stalling and template switching/microhomology-mediated break induced repair(FoSTeS/MMBIR)이다. |
| − | |||
| − | |||
| − | + | 두 번째는, '''암 유형에 따라 체세포 구조 변이를 확인'''하는 것이다. 14명의 직장암, 7명의 다중 흑색종, 7명의 전립선암, 9명의 심각한 난소 낭종암, 16명의 다형성 교모세포종, 19명의 간암, 35명의 침습적 유방암, 19명의 폐암, 10명의 자궁내막암, 4명의 신장암 환자를 포함하여 총 140명의 암 환자들에서 전장 게놈 해독 데이터를 분석하였다. 그 결과 25,874개의 구조변이를 확인하였고, 평균 185개 변이를 확인할 수 있었다. 암 유형에 따라 개인간에도 구조변이 수는 차이를 보였는데, ANOVA 도구(분산분석, 다수의 집단을 비교하고자 할 때 집단 내의 분산을 비교하는 것)를 이용하여 확인하였다. | |
| − | |||
| − | |||
| − | | + | 세 번째는, '''체세포 구조변이를 위한 돌연변이 메커니즘을 확인'''하는 것이다. 결실은 평균 46개 일어났는데, 대부분 DNA 두 가닥이 모두 중단된 것으로, 위 그림과 같이 예측이 가능하다. 암 게놈에서는 nonhomologous end joining (NHEJ)가 39%, alternative end joining (alt-EJ)가 41%로 높은 비율로 나타난다. 생식세포에서는 transposable element insertion (TEI) 변이가 높게 나타난다. 따라서 체세포의 중복과 복잡한 결실로부터, 짧은 염기서열이 같은 microhomology나 homology가 없는 non-homology가 일어나지 않는 template-switching 메커니즘을 추측하였다. |
| − | |||
| − | |||
| − | + | 네 번째는, '''다형성 교모세포종 환자에서 복잡한 염색체 재배열이 일어났음을 확인'''하는 것이다. 16명의 환자 데이터에서 이형의 염색체 10, 12번에서 CDKN2A/B의 동형 결실이, 14명에서 EGFR amplification이 발견되었다. 26개 구조변이를 검사해보니, PCR에 의해 체세포 구조변이로 25개에서 CDKN2A/B, EGFR, CDK4의 기능에 이상이 생겼다. | |
| − | |||
| − | |||
| − | + | 다섯 번째는, '''복잡한 재배열과 Double minute chromosome를 확인'''하는 것이다. Double minute chromosomes(DM)는 염색체 DNA 내에 centromere와 telomere가 자가 복제하지 않는 것이다. 백혈병과 같은 고형암의 변이에서 발견할 수 있다. | |
| − | |||
| − | |||
| − | + | === 맺음말 === | |
| − | |||
| − | + | ||
| + | 개인의 다른 암 유형에 따라, 진화 메커니즘과 체세포 구조변이 유형의 다양성을 유추하고, 암에서 체세포 재배열을 확인할 수 있었다. 예전에는 발현되지 않는 게놈의 기능 유전자는 연구되지 않았으나, ENCODE 프로젝트를 통해 최근에 이러한 게놈의 유전자들도 기능을 한다고 밝혀졌다. 다형성 교모세포종 환자를 통해 CDKN2A/B와 EGFR의 기능 변화를 통해 같은 게놈 내에서 같은 유전자리(locus)에서 동시에 발생하는 메커니즘을 발견했다. | ||
| + | |||
| + | |||
| + | |||
| + | === 참고논문 === | ||
| + | |||
| + | |||
| + | |||
| + | Diverse Mechanisms of Somatic Structural Variations in Human Cancer Genomes | ||
| + | |||
| + | |||
| + | |||
| + | === 역저자 === | ||
| + | |||
| + | |||
| + | |||
| + | 글 : Park.HyeonJi | ||
| + | |||
| + | 편집 : Jeon.EunSook, Ahn.Kung | ||
Latest revision as of 02:18, 11 October 2022
암 게놈에서 체세포 구조 변이의 다양성 메커니즘
암 게놈에서 체세포 변이 식별이 NGS 분석을 통해 가속화되고 있다. 그러나 구조 변이의 복잡한 특성을 알기에는 기본 메커니즘으로는 불충분하다. 생명의학 정보 센터와 하버드 의과대학 연구진들은 140명의 종양 환자들의 전장 게놈을 해독하여 체세포 변이를 예측하는 알고리즘에 적용하였다. 유전자 재배열과 같은 구조 변이들에 관여하는 돌연변이 메커니즘을 발견하였고, 이를 이용하면 염기서열 상에서 삽입과 결실, 복제 등의 차이를 규명할 수 있다. 특히, CDKN2A/B 유전자에서 염기 손실과 아교모세포종에 관련된 EGFR 복제에 대한 메커니즘을 확인하였다.
암은 단일 염기 변이(single nucleotide variation), 구조변이(structural variation), 염색체 이수성(aneuploidy) 등의 유전적 변화에 의해 발발한다. 특정 암 유형은 화학약물, 자외선, 흡연과 같은 것에 노출되어 특정 돌연변이와 복원 메커니즘이 고장 나서 발생하는 체세포 단일 염기 변이에 의해 분류된다. 큰 범위의 구조변이는 염색체 deletion, insertion, inversion, tandem duplication, translocation, complex rearrangement 등으로 인해 암에서 정상 유전자의 기능에 영향을 끼친다. 하지만 여전히 구조변이에 대해서는 밝혀진 것이 없다.
체세포 구조변이를 식별하는 5가지 방법
첫 번째는, Meerkat을 이용하여 생식세포의 복잡한 결실을 규명하였다. 암에서 체세포 구조변이 돌연변이의 특징은 복잡하고 간단한 구조변이를 모두 규명할 수 있다는 것이다. Meerkat이라는 새로운 알고리즘을 이용하면, Soft-clipped과 매핑되지 않은 부분을 확인할 수 있고, discordant read도 확인할 수 있다. 따라서 특이하지 않은 부위의 매핑 read들을 이용하여 조정하거나 복잡한 것은 짝을 이뤄 클러스터 시킬 수 있다. 서열이 중단된 지역을 이용하여 염색체 중단이 일어난 지역을 유추하고, alignment하여 메커니즘을 불러 올 수 있는 것이다.
이에 따라 6가지 분석 파이프라인으로 분류할 수 있는데, transposable element insertion (TEI), variable number of tandem repeats (VNTR), nonhomologous end joining (NHEJ), alternative end joining (alt-EJ), nonallelic homologous recombination (NAHR), fork stalling and template switching/microhomology-mediated break induced repair(FoSTeS/MMBIR)이다.
두 번째는, 암 유형에 따라 체세포 구조 변이를 확인하는 것이다. 14명의 직장암, 7명의 다중 흑색종, 7명의 전립선암, 9명의 심각한 난소 낭종암, 16명의 다형성 교모세포종, 19명의 간암, 35명의 침습적 유방암, 19명의 폐암, 10명의 자궁내막암, 4명의 신장암 환자를 포함하여 총 140명의 암 환자들에서 전장 게놈 해독 데이터를 분석하였다. 그 결과 25,874개의 구조변이를 확인하였고, 평균 185개 변이를 확인할 수 있었다. 암 유형에 따라 개인간에도 구조변이 수는 차이를 보였는데, ANOVA 도구(분산분석, 다수의 집단을 비교하고자 할 때 집단 내의 분산을 비교하는 것)를 이용하여 확인하였다.
세 번째는, 체세포 구조변이를 위한 돌연변이 메커니즘을 확인하는 것이다. 결실은 평균 46개 일어났는데, 대부분 DNA 두 가닥이 모두 중단된 것으로, 위 그림과 같이 예측이 가능하다. 암 게놈에서는 nonhomologous end joining (NHEJ)가 39%, alternative end joining (alt-EJ)가 41%로 높은 비율로 나타난다. 생식세포에서는 transposable element insertion (TEI) 변이가 높게 나타난다. 따라서 체세포의 중복과 복잡한 결실로부터, 짧은 염기서열이 같은 microhomology나 homology가 없는 non-homology가 일어나지 않는 template-switching 메커니즘을 추측하였다.
네 번째는, 다형성 교모세포종 환자에서 복잡한 염색체 재배열이 일어났음을 확인하는 것이다. 16명의 환자 데이터에서 이형의 염색체 10, 12번에서 CDKN2A/B의 동형 결실이, 14명에서 EGFR amplification이 발견되었다. 26개 구조변이를 검사해보니, PCR에 의해 체세포 구조변이로 25개에서 CDKN2A/B, EGFR, CDK4의 기능에 이상이 생겼다.
다섯 번째는, 복잡한 재배열과 Double minute chromosome를 확인하는 것이다. Double minute chromosomes(DM)는 염색체 DNA 내에 centromere와 telomere가 자가 복제하지 않는 것이다. 백혈병과 같은 고형암의 변이에서 발견할 수 있다.
맺음말
개인의 다른 암 유형에 따라, 진화 메커니즘과 체세포 구조변이 유형의 다양성을 유추하고, 암에서 체세포 재배열을 확인할 수 있었다. 예전에는 발현되지 않는 게놈의 기능 유전자는 연구되지 않았으나, ENCODE 프로젝트를 통해 최근에 이러한 게놈의 유전자들도 기능을 한다고 밝혀졌다. 다형성 교모세포종 환자를 통해 CDKN2A/B와 EGFR의 기능 변화를 통해 같은 게놈 내에서 같은 유전자리(locus)에서 동시에 발생하는 메커니즘을 발견했다.
참고논문
Diverse Mechanisms of Somatic Structural Variations in Human Cancer Genomes
역저자
글 : Park.HyeonJi
편집 : Jeon.EunSook, Ahn.Kung