From PGI
Contents
유방.전립선암에 대한 희귀 기능변이 추정 결과
현재까지 질병특성들과 유전적 연관성에 대해서는 아직 정확하게 밝혀지지 않은 것이 많다. GWAS(게놈 전체 연관성 연구)는 질병에 대한 위험이 있는 유전자 변이들의 식별에는 성공하였지만, 그 상대적 위험도를 설명하기에는 부족했었다. 그 설명의 수준은 특정 변이를 가지고 있으면, 질병이 유전되어 가족에게 위험이 있을 수 있다는 것의 일부만을 설명 할 수 있었다.
최근에는 희귀 단백질생산(coding)영역의 변이는 특정 질환에 복잡한 영향을 미친다는 가설을 선호하는 추세이다. 유방암과 전립선 암의 많은 위험변이도 GWAS를 통하여 이미 많은 수(유방암, 전립선암의 경우 70개 이상)가 밝혀졌지만, 밝혀진 모든 변이들은 일반적인 변이로, 소수의 위험만을 설명할 수 있다. 대부분의 GWAS는 특정 인구 내의 단백질생산(coding) 및 단백질비생산(non-coding) 영역에서 일반적인 큰 변이를 찾아내는 데, HapMap('Haploid'와'Map'의 합성어 : 포스게놈 시대의 새로운 연구 분야로 떠오르고 있는 반쪽짜리 게놈지도(유전자지도)를 말한다. 합맵프로젝트 라고도 한다.유전자 정보의 일부만을 밝히는 데 중점을 둔다는 뜻에서 이런 이름이 붙었다.) 에서 선택된 SNP(Single Nucleotide Polymorphism)와 llumina와 Affymetrix의 SNP 배열을 사용했다. 이런 방법으로 얻어진 데이터 뿐 아니라, 낮은 스펙트럼을 보이는 유전자 변이도 인간 게놈에 있는 모든 변이에서 큰 부분을 차지하기 때문에, 어떤 유전변이가 어떻게 질병의 원인이 되는지에 대해서는 아직 모두 밝혀지지 않은 것이다.
매우 희귀하거나 얕은 침투도를 가진 변이가 질병 유전에 역할을 한다는 가설을 증명하려면 그 변이들의 발견을 위해서는 매우 큰 샘플 크기의 연구를 필요로 한다. 최근 기술에서는 이러한 큰 샘플 사이즈의 연구에서 분석 부담을 줄이기 위한, 통계지원이 제공되고 있으며 정확히 필터 된 희귀 변이들을 탐색한다.
본 연구에서는 개인의 필터 된 배열 순서뿐만 아니라 모든 희귀변이의 배열도 알 수 있는 Illumina Infinium Human Exome Array 를 사용하여, 코딩 영역에서의 희귀한 변이가 다민족인구(아프리카계 미국인, 라틴계 미국인, 일본계 미국인, 하와이 원주민, 유럽계 미국인)의 암 위험도에 기여한다는 가설을 환자군(유방암, 전립선 암 환자) 및 정상인 샘플에서 분석했다. 이번 연구의 의의는 일반적으로 중요한 역할을 하지 않는다고 생각하는 단백질생산(coding) 영역의 변이들 중, 위험 유전에 확연한 역할을 하는 변이에 대해 알아보는 데에 두었다.
다민족 연구사례 특징
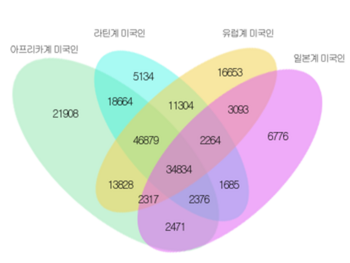
이번 연구에 참가한 다민족은 캘리포니아와 하와이에 거주하는 다섯 개의 민족/인종(아프리카계 미국인, 일본계 미국인, 라틴계 미국인, 하와이 원주민, 유럽계 미국인)의 45-75세로 구성하였으며, 총 7,815명의 환자(3,141명의 유방암 환자, 4,674명의 전립선암 환자)와 8,021명의 정상인을 대상으로 하였다. 위 그림은 하와이 원주민을 제외한 4개 민족/인종 그룹의 공유변이 개수를 추정 그림이다. (하와이 원주민에서는 공유된 변이수가 적어 제외되었다.)
유방암 환자의 경우 에스트로겐 수용체 양성 종양(ER+, 전체 유방종양 중 약 70~75% 차지)과 음성 종양(ER-, 전체 유방종양 중 25~30% 차지)으로 분류하였다. 전립선암은 전이성 종양과 비전이성 종양으로 분류하였다. 해당 대상자들은 이전의 건강 상태 등의 내용이 포함된 설문 정보를 추가하였다.
지노타이핑과 SNP 분석
지노타이핑은 USC(university of southern california) 유전체학 코어 실험실에서 Infinium Human Exome BeadChip을 이용하여 진행하였다. 환자와 정상인은 암 유형 별 인종 분류에 따라 칩에 배치했으며, Genome Studio 소프트웨어(V.2011.1)를 사용하여, 확인된 모든 결과를 자동 클러스터링 하였다. 그리고 하디-와인버그 평형을 거쳐, 친척관계로 추정되는 샘플을 제거하였다. 하디-와인버그 평형이란, 유전적 구성이 일정한 집단에서 이루어지는 유전자빈도의 평형 모델을 말하며, 임의교배가 이루어지는 이상적 집단에서 선택, 돌연변이, 이주 및 유전자 부동이 없는 경우 집단 내 유전자 빈도는 세대가 바뀌어도 일정 수준에서 평형을 유지하고 보전한다는 이론을 사용한다. 이러한 과정을 거쳐 2,984명의 유방암 환자와 3,568명 정상인, 4,376명의 전립선 환자와 3,977명의 정상인의 245,339 SNP로 분석하였다. 그 결과, 다민족 샘플 중 88%의 기능변이가 발견되었고, 그 중 12%에서는 두 개의 희귀한 변이가 발견되었다. 단일변이 비율은 아프리카계 미국인 34.1%, 유럽계미국인 39.6%, 하와이 원주민 66.8%, 일본계 미국인 74.2%, 라틴계 아메리카인 43.4%로 나타났다.
통계적 분석은 기능변이의 추정에 초점을 맞추어 진행되었으며, EIGENSTRAT를 사용하여 전체 샘플에서 상위 10개 유전자 변이를 추론하였다. EIGENSTRAT sofrwere는 GWAS에서 인구 층화에 대한 수정 프로그램으로, 모델의 변화의 연속축을 분석하여 케이스와 컨트롤의 조상차이를 알아내는 분석법이다. 이 방법으로 진정한 연관을 감지하고, 거짓 연관을 최소화 시킬 수 있다. 정확도를 위해 정상인은 성 염색체를 제외하고 분석하였으며, 같은 성별간의 비교에서 성 염색체를 포함하여 분석하였다. 상위 10개 유전자에 대해서는 로지스틱 회귀분석법을 실시하여 기존에 알려져 있던 유전자 위치와 근처에서 발견된 새로운 변이를 확인하였다. 로지스틱 회귀분석은 분석하고자 하는 대상들이 두 집단 혹은 그 이상의 집단으로 나누어진 경우에 개별 관측치들이 어느 집단으로 분류될 수 있는가를 분석하고 이를 예측하는 모형을 개발하는 데에 사용되는 대표적인 통계기법이다.
많은 인종과 민족 다형성을 고려하여 본페로니(Bonferroni) 판정법을 이용하였다. 본페로니 판정법이란, multiple testing의 문제를 해결하는 가장 기본적인 방법으로서, 개별 가설 검정을 일정 유의 수준(특정 유전자의 단일 변이 수에 따라 달라짐)으로 검증한다. 이 방법의 장점은 에러를 5% 이내로 조절하는 데 있으며, 본페로니 판정법의 결과가 유의하면, 다른 방법에서도 유의한 결과를 나타낸다.
유방암, 전립선암의 위험과 연관된 SNP
유방암의 경우, 인종 별 분석에서 가장 중요할 것으로 추정되는 유전자는 거의 독점적으로 아프리카계 미국인에게서 관찰되는 LDLRAD1로 밝혀졌다. 나머지 9개의 연관 NS(nonsynonymous)변이들은 본페로니 판정법을 통과하지 못하였다.
각 유전자의 모든 단일 변이를 기반으로 본페로니 검증을 거친 연구결과에서는 MMAB, SLC16A6, INS-IGF2 유전자에서 강한 연관성이 관찰되었다. MMAB유전자는 HDL 콜레스테롤과 연관된 단일 변이 위치와 가까웠다. SLC16A6 유전자는 monocarbohydrate 수송자(monocarbohydrate transporter)이다. INS-IGF2 유전자는 인트론에 위치하며, 1형 당뇨병과의 연관이 밝혀진 바 있었다.
전립선암과 관련된 F13A1유전자도 이전에 보고된 적은 없었으나, 이번 연구에서 확인되었다. ANXA4유전자는 위암에 연관된 표지자 였으며, GPRC6A유전자는 NS변이로 분류되었던 표지자였다. 하지만 3가지 전립선 암과 연관된 유전자로 발견된 유전자들은 로지스틱 회귀분석을 거친 결과에서 높은 P값을 보였으며, 본페로니 판정법에서도 유의하지 않아, 결과적으로 인종 별 분석에서 전립선암과의 연관성이 크지 않다고 판단되었다.
하지만 유방암과 연관된 것으로 밝혀진 LDLRAD1 유전자의 경우처럼, 기존에는 제외되었던 낮은 빈도의 변이들도 특정 질병과의 상당한 연관성을 알 수 있었다. 이전에는 유방암이나 전립선암과의 연관성에 대해 보고된 적이 없었으나, 이번 연구를 통해 새로이 확인된 것이다.
맺음말
단백질생산(coding) 영역에서의 희귀 변이와 질병과의 유전률에 대해 추정한 이번 연구에서는, llumina Human Exome SNP array를 사용하여 다민족에서 성공적으로 지노타이핑 하였다. 그 중에서는 기존에 유방암과 연관성이 높은 단일 변이와 유전자도 포함되어 있었고, 전립선암은 알려진 유전자 근처에 위치한 새로운 유전자를 발굴하였다. 연관된 유전자들의 대부분은 이전에 밝혀졌던 암에 관련된 유전자와의 연관성이 있거나, 다중보정 후 탈락되었다. 그러나 LDLRAD1유전자의 경우는 유방암 연관으로는 밝혀진 적이 없던 유전자로서, 다중보정 후에도 통계적으로 유의하였다. 이로써 유방암에 관련된 새로운 잠재적 유전자로 설명할 수 있었다.
유방암과 전립선암에서 높은 위험성의 단백질 코딩 변이들은 odds ratio(=오즈비 : 사례 대조연구에 있어서 상대위기(relative risk)의 추정치로서 산출된다.) 범위를 넘어서기 때문에,연구 결과로서는 제공되지 않았다. 이후 연구들은 샘플의 범위를 넓게 하여 희귀 변이의 역할에 대해 재조명하게 될 것이다.
참고문헌
Genome-Wide Testing of Putative Functional Exonic Variants in Relationship with Breast and Prostate Cancer Risk in a Multiethnic Population
http://www.plosgenetics.org/article/info%3Adoi%2F10.1371%2Fjournal.pgen.1003419
저자
글 : Jung.EunByoul
편집 : Park.HyeonJi, Jong.Bhak
키워드 : LDLRAD1, GWAS, SNP(Single Nucleotide Polymorphism), HapMap, llumina Human Exome SNP array, Bonferronie 등