From PGI
Contents
1,092명의 인간 게놈 변이지도를 통합해내다!
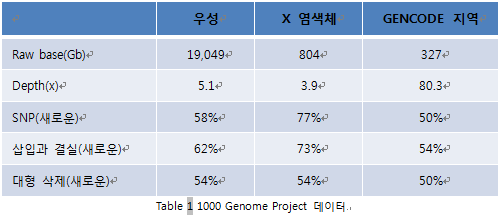
인간의 유전적 변이의 위치, 기능적 특징을 분석하는 일은 오랜 숙원이였다. 최근 질병에 대한 유전적 기여를 이해하기 위해, 1000 게놈 프로젝트 컨소시엄에서1,092명의 개인의 전장유전체 및 엑솜을 시퀀싱한 결과가 보고되었다. 본 연구는 1%의 개개인의 특징 중 지금껏 연구하지 않았던 낮은 빈도의 희귀한 생물학적 변이들을 분석하였다. 3800만개 SNP(Single Nucleotide Polymorphism)와 indel 140만개, 삭제된 1만4천여개를 haplotype의 지도로 표현하였다.
1000 Genome Project1)
1000 게놈 프로젝트는 유전자형과 표현형 사이의 관계를 조사를 위한 기초로 인간 게놈 서열의 특징을 제공하는 것을 목표로, 7가지 인종에서 697명의 엑솜시퀀싱과 4가지 인종에서 179명의 전장유전체 시퀀싱을 NGS 플랫폼(Illumina)으로 연구하였다.
유전자의 위치, 대립유전자 빈도와 약 1500개 SNP, 100만개 indel, 그리고 2만개의 구조적 변이를 포함한 haplotype구조를 설명한다. 평균적으로, 유전자의 약 250-300개 기능이 손실되는 변이가 발견되었다. 이 결과는 GWAS(Genome wide association study)2) 연구에서 trio 분석을 통해 증명하였고, 자연적인 선택에 대한 데이터를 수집하였다.
유전체에서 SNP의 많은 구조적 변형으로 변이가 낮은 것에는 관심이 없었으나, 잠재적으로 기능의 변이에 대한 풍부한 예로 작용할 것이라는 추측으로 연구를 진행하였다. 결과적으로 인종 다양화의 수준을 증가시키며 포인트변이3) 및 구조의 변형, 중요한 기능에 많은 변이를 식별할 수 있었다. 본 연구에는 유럽, 동아시아, 사하라 사막 이남의 아프리카와 아메리카 등 다양한 인종을 대상으로 하였으며, 3,415개의 대형 삭제된 부위부터 281개의 낮은 부위와 185개의 엑솜 위치까지 결과를 포함하였다.
전장유전체 시퀀싱으로 99.3%을 읽은 후 엑솜시퀀싱을 추가하여 99.8%를 읽었다. SNP와 indel, SV(Single Variant)를 포함하여 정확도를 높였다. 유전적 다양성을 분석하고, haplotype의 정확성을 확인하기 위해 어머니와 아버지, 자손의 trio에서 수집한 SNP 데이터를 비교하였다. 본 프로젝트의 목표는 광범위한 인구 집합의 1% 빈도에서 SNP 95%를 파악하는 것이었다. SNP와 indel 사이트의 낮은 빈도의 복잡한 유전체 지역의 변이를 확인하였다.
인종간4) 유전적 변이의 차이
인종간 유전자의 변이 차이를 보기 위해 특정 질병에 원인 유전자로 알려진 변이 부위를 분석하였다. 예를 들어, 신장 질환에 관련된 ALMS1, NAT8 유전자의 인종 별 변이를 조사하였다. 하늘색은 참조 대립 유전자이고, 분홍색은 고밀도 SNP 부위이며, 흰색은 이전에 알려진 변이들, 진한 파란색은 새로운 변이들이다. 예를 들어, 유럽인 중 IBS와 FIN 인종의 경우 Kb당 대립 유전자의 빈도가 높게 나타났다. 또한 파생되는 대립 유전자의 빈도 분포는 상당한 아프리카의 조상 인구에서 다양한 변이가 발견되었다. 단일 인종의 희귀 변이를 확인하기 위해 무작위 샘플(흰색)과 비교하였다. 모든 인종에서 최근 폭발적인 인구의 증가와 희귀 변종에 대한 차별적인 반응을 볼 수 있다.
희귀한 변이 중 모든 인종에서 나타나는 변이들을 표시하고, 연령에 따른 한 가닥의 대립 유전자 길이는 반비례하며, 물리적 유전적 거리에 대해 GBR 인종으로 확인하였다. 또한 1Mb 이상의 거리에서는 부분적 대립유전자를 공유한다는 사실을 밝혀냈다. 희귀한 SNP 주변 haplotype에 대해 아메리카 원주민(NatAm)으로 비교하여, 서로 다른 조상에서 변이 발생의 평균 비율을 확인하였다. 역사적 혼혈과 인구의 다양성을 분석하기 위해, 인종 다양성의 대표적인 국가인 미국인 샘플에서 다양한 조상 지역의 유전적 다양성에 대해 조사하였다. 아래 그림에서 푸른 색은 유럽계, 갈색은 아프리카계, 붉은 색은 미국 원주민, 검은색은 밝혀지지 않은 부위이다.
기능적 변이 스펙트럼
두 인종의 희귀 변이의 비율과 진화와 보전 사이의 연관성에 대한 분석 결과이다. 기능 유전자의 변이 유형과 진화와 보존 사이의 대립 유전자 분포 빈도와 인종 사이의 차별화를 조사하였고, 유전자 집합에서 희귀한 해로운 돌연변이로 인해 발생한다는 사실을 확인하였다. KEGG5) 경로의 유전자 그룹간 분리가 이루어져 희귀한 분화를 이루었다. 암호화된 유전체의 기능 다형성에 대한 정보를 제공한 이번 결과는 높은 보존력을 가진 CTCF 단백질 결합으로 인한 전사를 억제하는 결과도 포함하였다. ChIP-seq을 통해 CTCF에 결합하여 높아진 봉우리는 진화에서 보존되는 모티프로 정의되었다.
진화적으로 보존되는 부위들을 GERP 점수로 나타내었고, 대립 유전자에서 파생되었는지 연관성을 표시한 것이다. ChIP-seq의 결과로 밝혀진 CpG 위치와 피크(빨간색)를 통해 대립유전자의 위치를 파악하고 ENCODE6)에서 나온 결과(파란색)와 비교하였다.
유전의학에서 게놈 변이를 이용할 수 있을까?
유전 질환과 암 환자의 엑솜 데이터에서 발견된 변이들은 진화과정에서 보존되어야 할 부위의 개인 변이들이다. 2,500개의 이형 변이들 중 150개 기능 손실 변이들은 암 환자의 유전체 시퀀싱에서 유해 변이로 이전 연구에서 확인되었다. 이번 연구에서는 76~190가지 희귀 이형 변이 및 최대 20가지의 기능 손실 변이 및 질병에 관련된 변이를 발견하였다. 질병 후보 유전자를 식별할 때는 변이의 빈도를 고려하는 반면, 이번 연구에서는 낮은 빈도의 변이들에 대해 연구하였기 때문에 조절 기능에 대한 정보에 초점을 두고 진행되었다. 변이 데이터의 결합 가능성이 비 암호화된 변이를 감지하고 유전자의 조절과 기능에 약하지만 해로운 영향을 미칠 가능성을 염두하였다. 26가지 유전형 정보를 연구한 GWAS(Genome wide association study)를 대상으로 GWAS 연구에 분석할 종류에 따른 변이 빈도를, 분석 방식에 따른 SNP간 거리를 감지하였다.
GWAS 연구로 광범위한 haplotype 구조의 단일 변이에 매핑한 예를 소개하였다. 56개 변이와 유전자 간 거리가 불균형하였다.
맺음말
멘델리안 질병의 유전적 복잡한 질환과 관련된 유전자의 희귀한 질병에 변이의 발견에 엑솜시퀀싱은 강력한 가설을 지원한다.GWAS 연구의 해석에 도움이 될 뿐만 아니라 프로젝트뿐만 아니라, 질병의 시퀀싱 기반 연구를 분석하는 방법에 대한 수업을 제공하였다. 단일 변종의 약 60%가 엑솜 데이터에서 발견되었다. 유전적 변이, 후보 변이 등의 품질을 평가하기 위해 통계적 방법으로 haplotype 통합하였다. 또한 많은 대규모 구조 변이 위치에 대해 조사하고, 특정 질병이 있는 사람들의 희귀 변이는 유전적 연관성 내에서 시행되었다. 다양한 인종에서 개인의 서열 가치에 대해 다시 한 번 생각해 볼 수 있는 연구가 된 것이다.
- 1000 Genome Project : 대용량 데이터들이 순차적으로 대량의 인간 게놈 데이터가 공개, 다국적 연구자들이 진행하고 있는 1000 Genome Project는 인간의 유전적 다양성에 대한 보다 엄밀한 청사진을 그리는 것을 목표로 하는 계획으로 제2의 HGP라 할 수 있다.
- GWAS(Genome wide association study) : 전 게놈 관련분석(Genome-wide association study; GWAS)은 병질환 및 약물 반응성에 대한 유전적 요인을 총체적으로 탐색하는 연구 방법을 말하며, 일본 이화학연구소의 Ozaki 그룹에서 최초로 시도된 연구 방법
- 포인트 변이(point mutation) : 하나의 뉴클레오티드가 다른 것으로 치환됨으로 발생하는 돌연변이.
- 인종 : SW(African ancestry in Southwest United States), CEU(Utah residents with ancestry from Northern and Western Europe), CHB(Han Chinese in Beijing, China), CHS(Han Chinese South, China), CLM(Colombians in Medellin, Colombia), FIN(Finnish in Finland), GBR(British from England and Scotland UK), IBS(Iberian populations in Spain), LWK(Luhya in Webuye, Kenya), JPT(Japanese in Tokyo, Japan), MXL(people with Mexican ancestry in Los Angeles, California), PUR(Puerto Ricans in Puerto Rico), TSI(Toscani in Italia), YRI(Yoruba in Ibadan, Nigeria Ancestry-based groups), AFR(African), AMR(Americas), EAS(East Asian), EUR(European)
- KEGG(Kyoto Encyclopedia of Genes and Genomes) : 1995년 일본에서 만들어진 생화학 pathway 관련 데이터베이스이다. 일본에서 만들어진 DB중에는 세계에서 가장 많이 인용되는 것의 하나에 속한다.
- ENCODE project : 인간 게놈 안의 모든 기능적인 요소를 동정하겠다는 목표로 전사물이나 유전자 간 관계를 규명하는 프로젝트
참고문헌
An integrated map of genetic variation from 1,092 human genomes
http://www.nature.com/doifinder/10.1038/nature11632
Evolution and functional impact of rare coding variation from deep sequencing of human exomes.
http://www.sciencemag.org/content/337/6090/64.abstract
An abundance of rare functional variants in 202 drug target genes sequenced in 14,002 people.
http://www.ncbi.nlm.nih.gov/pubmed/22604722
저자
글 : hjpark
편집 : Thkim
글 오류 신고는 hyeonji.park@pgi.re.kr으로 보내주세요.
키워드 : 1000 Genome Project, GWAS(Genome wide association study), haplotype, SNP, indel, KEGG(Kyoto Encyclopedia of Genes and Genomes), ENCODE project 등