From PGI
Contents
운남성 염소의 모낭은 뭔가 다르다?
염소는 10,000년 전 야생종을 사육하기 시작하면서 현재는 1,000여종 이상의 품종이 전 세계적으로 8억 3천여 마리가 자생하고 있다. 염소는 고기, 우유, 섬유, 가죽 등으로 이용되며 농업, 경제, 문화, 종교 등 다방면에서 활용되고 있다. 염소는 가축으로서의 가치도 있지만, 약물의 변이된 유전자를 생산하는 의학 연구 동물 모델로서의 가치도 있다. 그러나, 사육 및 유전학 연구에 참조 유전체 서열의 다양성이 부족하다.
이에 중국 쿤밍의 유전 진화 연구소에서는 Capra hircus 종의 유전체를 최초로 해독(de novo sequencing)하여 복잡한 특성의 유전 기초를 조사하였다.Capra hircus 종은 중국, 인도를 비롯한 기타 개발도상국에서 사육되어왔다. 중국 운남성(윈난성으로 불리기도 함)의 염소(Capra hircus)의 전장 유전체 해독과 fosmid 해독을 통해 2.66Gb의 유전체를 확인하였고, 소와의 공통 조상에서 보존된 유전자의 synteny(염색체 상의 유전자 구성의 상동성)에 따라 염색체에서의 위치를 확인하였다. 또한 10가지 조직을 전사체 분석(RNA-Seq)하여 22,175개 단백질 코딩 유전자를 확인하였고, 모낭의 두 가지 유형의 발현에 관련된 51개 조절 유전자에 대해 살펴보았다.
이번 연구는Illumina Genome Analyzer II를 이용한 NGS 방법을 이용하였고, 대형 DNA를 매핑하는 방식(whole genome mapping)을 이용하여, 고품질의 유전체 지도를 완성하였다. 이전의 박테리아 유전체 어셈블리로 구조를 비교하는 Sanger sequencing(생어시퀀싱) 방법은 복잡한 유전체 어셈블리를 수행하지 못하는 한계점이 있지만, NGS를 이용하면 비용과 시간이 절감되며, 복잡한 구조도 해독이 가능하다.
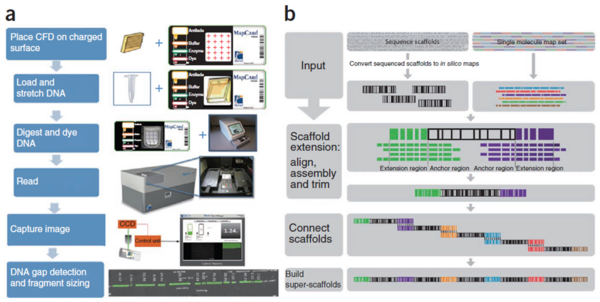
그림 1 whole genome mapping 연구 흐름도
[그림 1]은 whole genome mapping 방법의 연구 흐름도 이다. [그림 1]의 a는 샘플을 칩에 로딩시켜 높은 밀도의 채널 형성 장치(CFD; channel-forming device)에 걸어 DNA gap을 감지하고 조각의 크기를 확인하는 방법의 워크플로우이다. [그림 1]의 b는 NGS와 Optical mapping system을 이용하여 de novo 어셈블리 데이터를 두 가지 제한 효소로 잘라 하이브리드 어셈블리하는 방법의 워크플로우이다.
염소의 유전체 해독 및 어셈블리
그림 2 간략한 연구흐름도
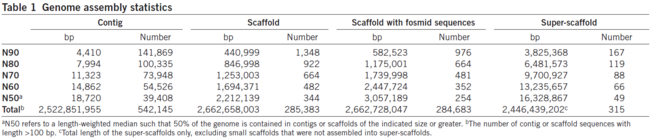
표 1 유전체 어셈블리 통계치
운남성에서 서식하는 염소 암컷(3세)의 간 조직에서 DNA를 추출하여 다양한 크기(180bp, 350bp, 800bp, 2kb, 5kb, 10kb, 20kb)의 라이브러리를 제작하여 Illumina paired end sequencing(191.5Gb)을 수행하였다. 어셈블리는 SOAPdenovo(v.1.03)으로 수행하였으며, [표 1]은 NGS 데이터를 이용하여 만든 de novo assembly 결과이다. contig와 scaffold의 N50 결과를 보면, contig에선 18.7kb인 반면, 다양한 점핑 라이브러리를 이용하여 얻은 scaffold에선 약 120배 가 긴 2.21Mb의 결과를 만들었다.
이번 연구에서는 염소의 고품질의 유전체 지도를 생산하기 위해 whole genome mapping 방법을 이용하였고, 전체 염색체 길이를 contig1)와 scaffold1), super-scaffold1)순서로 정렬하였다. NCBI에 등록된 다른 염소 종의 참조 유전체에 annotation하여 염소 유전체의 92%를 커버하였다. 포유류 6가지 모델의 핵심적인 진핵세포의 유전자 매핑 방식의 파이프라인을 이용하였더니, 97.58%의 유전체를 커버할 수 있었다.
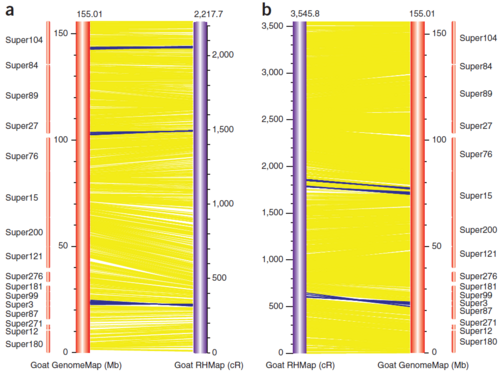
그림 3 super-scaffold와 방사선 하이브리드 지도(RHMap) 사이의 colinearity
염소는 상염색체 29쌍, 성염색체 1쌍(2n=60)으로 총 30쌍의 염색체을 보유하고 있으며, 소와의 세포유전학적 비교를 통해 1:1 대응관계가 존재하는 공직선성(colinearity)2)이 높게 나왔다. [그림 3]에서는 super-scaffold와 방사선 하이브리드 지도(RHMap3)) 사이의 공직선성을 확인할 수 있다. RHMap은 방사선을 이용하여 super-scaffold의 특정 마커 간의 거리를 물리적으로 확인하여 염색체의 작은 조각(contig, scaffold, super-scaffold)의 순서를 정해 재정렬한 지도이다.
[그림 3]의 a는 OvineSNP50 BeadChip으로 염색체 1번의 지도를 보여주는 것이며, b는 소 유전체와의 비교를 통한 염색체 1번의 지도를 보여주는 것이다. RHMap를 통해 완성된 염색체 지도에서 특정 염색체의 위치에 할당된 28개 염소 유전자를 확인하였다. 또한 X 염색체에 관련된 유전자 그룹은 태반 포유류에서 공통적으로 보존되지만, 소와 염소 사이에서는 유전자의 순서가 달랐다. 염소의 super-scaffold 정렬이 소 synteny4) 정보를 참조하지 않고 어셈블리 되어 순서가 크게 다른 것으로 나타났으며, 이를 통해 두 종의 분기를 예측할 수 있었다.
염소 유전체의 반복서열과 트랜스포존
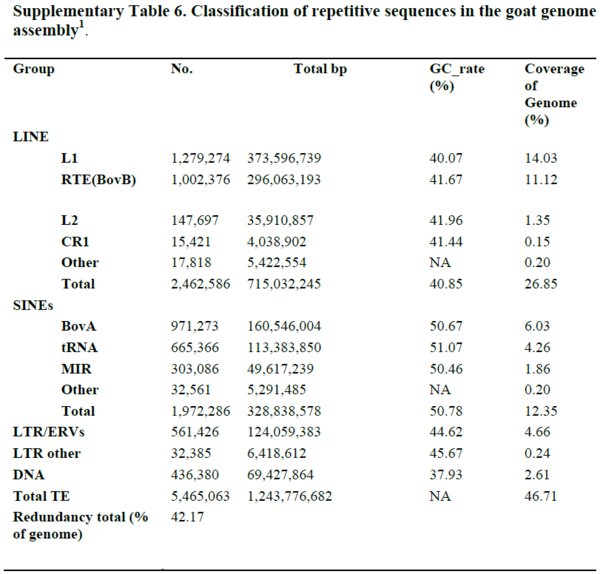
표 2 염소 유전체 어셈블리에서 반복서열 분류
트랜스포존(transposon)은 이동성 유전인자로 유전자와 유전체의 진화에 기여하는 요소이다. 염소 유전체의 42.2%가 이러한 트랜스포존으로 구성되어 있으며, 반추동물은 반복적인 서열의 트랜스포존이 대부분이라는 특징을 가지고 있다. 반추동물은 되새김동물이라고도 하는 4개의 위를 가지며, 반추위에는 미생물이 살고 있어 영양적 공생관계를 유지하는 소, 양, 사슴 등의 동물을 의미한다. 트랜스포존의 반복적 서열의 특징으로 반추동물과 반추동물이 아닌 동물의 차이를 확인할 수 있었다.
[표 2]는 염소 유전체 어셈블리에서 반복서열을 분류한 것으로, LINE는 RNA 중합효소 II에 의해 전사되거나 역전사된 RNA를 보여주는 긴 DNA배열이고, SINE는 RNA 중합효소 III에 의해 유래되거나 역전사된 RNA를 보여주는 짧은 DNA배열이다. 염소는 트랜스포존 서열의 80%이하가 SINE-BovA가 반복되었고, 소는 트랜스포존의 40%이상이 SINE-tRNA가 반복된 서열이었다.
염소 유전자 annotation
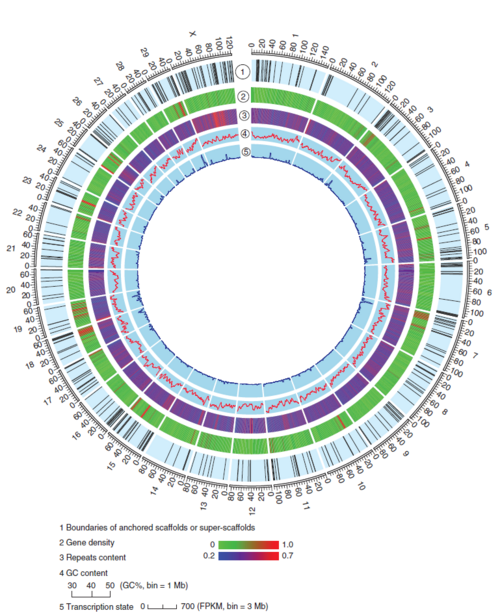
그림 4 염소 염색체 어셈블리 요약
Annotation을 이용한 유전자를 예측하는 방법은 3가지가 있다. 상동성을 기반으로 한 annotation, ab initio 예측, RNA-Seq/EST/tDNA 기반의 annotation이다. 이러한 방법을 이용하여 염소 유전체를 annotation하여 8개 exon당 유전자 1개 비율로 어셈블리된 것을 확인하였다. [그림 4]는 염소 유전체 어셈블리 결과를 이데오그램(ideogram)으로 나타낸 것으로, 이데오그램은 특정한 개념이나 의미를 대표하여 전달하는 데 사용되는 도형문자이다. 염소의 30쌍 염색체 길이, 유전자 밀도, 반복 서열, GC 컨텐츠 비율, 전사체 위치 등의 정보를 함유하고 있다.
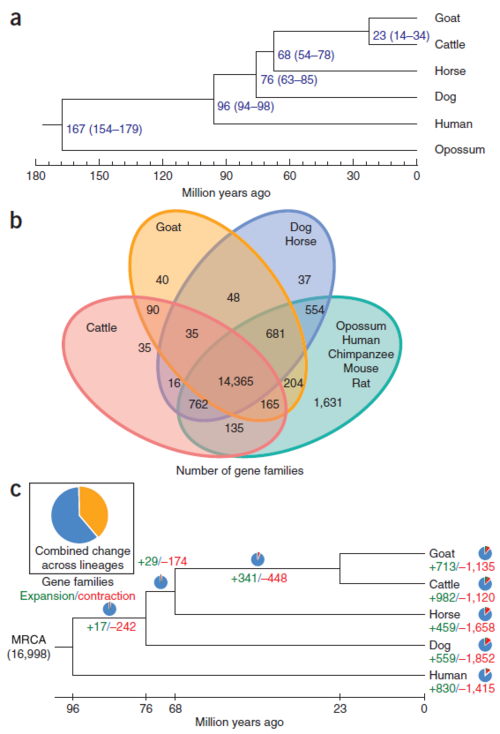
그림 5 계통분류 나무
[그림 5]는 이데오그램으로 확인된 정보로 계통분류 한 것이다. [그림 5]의 a는 4DTv(fourfold degenerate site)5) 방법으로 8,325개 단일 복제 유전자를 확인하여 분기시기와 독립적인 염기서열을 확인하였고, b는 9개 포유류 종의 유전자 가족 수를 벤 다이어그램으로 비교하였다. c는 유사 유전자 클러스터를 통해 공통 조상의 뿌리를 확인하였다.
이를 통해 염소와 소는 공통 조상에서 23만년 전 분기되었다는 것을 알 수 있다. 또한 염소는 면역 관련 유전자가 소보다 7개 많았고, 소와 유사한 유전자 44개가 급속도로 진화된 것을 밝혀냈다. 그 이유는 달라진 소의 유전자 클러스터가 우유 생산 및 털의 형태가 변화하는 등의 뇌하수체 호르몬에 관련되어 있었기 때문에 분기 이후 급속한 진화가 이루어진 것이다.
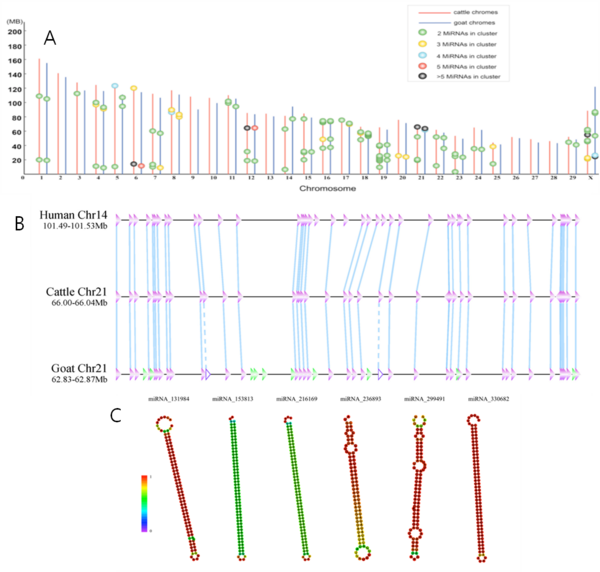
그림 6 miRNA 클러스터
[그림 6]은 염소의 miRNA 클러스터를 조사한 것이다. [그림 6]의 A는 염소와 소의 miRNA를 비교한 것이고, B는 사람과 소, 염소의 miRNA 클러스터와 synteny의 연관성을 나타낸 것이다. C는 염소의 miRNA와 인간, 소, 개, 침팬지, 쥐, 염소 등 9가지 포유류 종과 비교하여, 염소의 특이성 miRNA 6개를 나타낸 것이다.
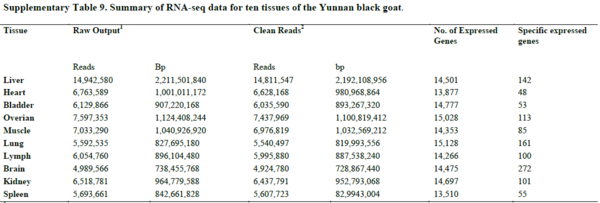
표 3 염소 10개 조직에서 RNA-Seq 결과
[표 3]에서는 염소의 10가지 조직의 전사체 분석을 정보를 나타낸 것이다. 염소는 소의 7배, 인간의 2배 많은 수의 후각 수용체 관련 유전자가 발견되었는데, 이는 염소의 뛰어난 채집, 수렵 능력에 기여를 한 것으로 보여진다. 그 중 하나인 FTH1 유전자는 간이나 비장에 존재하는 철분 저장 및 해독능력을 가진 ferritin 단백질을 발현시킨다. Ferritin으로 인해 염소는 식이조절 능력이 있다.
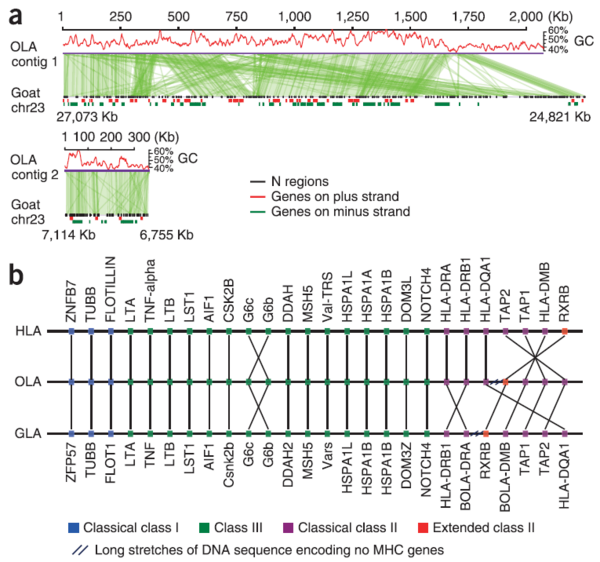
그림 7 염소의 MHC 비교분석
[그림 7]은 염소의 주조직 적합 복합체(Major Histocompatibility Complex, MHC)를 비교 분석한 것이다. MHC는 면역시스템에 관련 있는 유전자 군으로 세포표면에서 동종의 항원군에 대해 거부 반응을 하며, 유전적 다형현상이 특징적이다. [그림 7]의 a는 양과 염소의 MHC에서 1:1 비교이며, b는 유전자 보존지역에 한하여 사람, 양, 염소의 MHC를 비교하였다. 염소는 염색체 23번에 MHC loci가 위치하였으며, 따라서 23번 염색체는 면역 연구와 백신 개발에 도움이 되는 부위로 확인되었다.
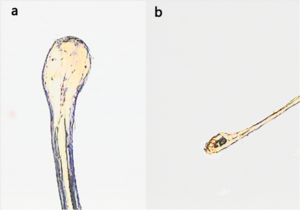
염소의 두 가지 모낭 형태
그림 8 염소의 두 가지 모낭 형태(PFH, SFH)
포유류의 모발은 피부 내 모낭에 의한 케라틴 조직으로, 일반적으로 두 가지 형태가 있다. 기본형 모발은 거친 코트의 털을 생산하는 형태이고, 보조형 모발은 캐시미어를 생산하는 형태이다. 원시 캐시미어는 중국에서 생산되기 시작하여 2,500년의 역사를 가졌으며, 매년 몇 만 톤씩 중국에서 생산된다. 이러한 캐시미어 섬유의 개발에 근간을 두고 유전자를 조사하였다.
캐시미어 세 가지 전사체 샘플을 준비하고, FPKM(fragment per kb exon model)에 따라 두 가지 모낭 형태를 분리하여 유전자 수를 확인하였다. FPKM은 전사체 단위에서의 발현 단위로 보면 되는데, 하나의 유전자에서도 여러 이형 전사체가 존재 가능하다는 특징이 있다. FPKM이 0.1이상인 경우 기본형 모발에 관련된 유전자가 10,077개이고, 보조형 모발에 관련된 유전자가 7,772개이다. FPKM이 5이상인 경우는 기본형 모발에 관련된 유전자가 2,572개이고, 보조형 모발에 관련된 유전자가 1,947개이다.
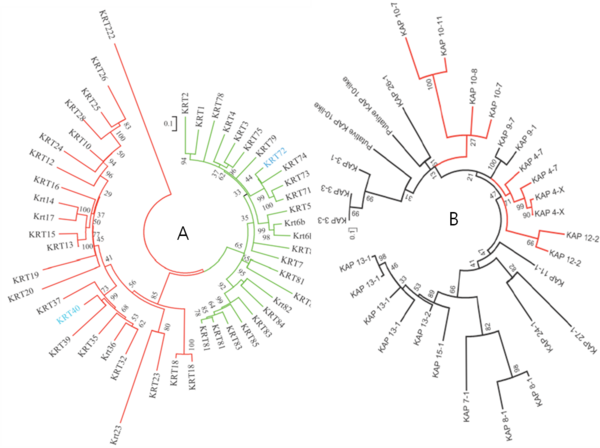
그림 9 염소 게놈에서 케라틴 발현 유전자 계통나무
모발의 발현을 조절하는 51개 유전자(감소 조절 28개, 증가 조절 23개)는 섬유 구조 단백질인 케라틴의 발현에 따라 섬유의 품질을 결정한다. [그림 8]은 모발의 발현을 조절하여 두 가지 모낭 형태로 발현된 결과이다. [그림 8]의 a는 기본형 모발, b는 보조형 모발이다. [그림 9]는 모발의 발현에 관련된 유전자의 계통을 보여주는 것으로, A는 케라틴 관련 유전자이고, B는 각질 관련 유전자인 KAP 유전자이다.
보조형 모낭의 경우, 증가 조절 유전자는 섬유아세포의 성장, 카세인의 Iε 효소로 인해, Wnt 신호 경로의 β-catenin이 조절되어 나타난다. 감소 조절 유전자는 아스파라긴과 세린 생합성 효소와 아미노 전이효소(phosphoserine)로 인해, Wnt 신호 경로가 조절되어 모발의 성장과 수명주기, 레티노산 결합 단백질의 발현이 이루어진다.
맺음말
염소와 같은 대규모 유전체에 대한 높은 연속성의 어셈블리를 얻기 위해 whole genome mapping 방법을 사용하였다. 완성된 유전체 지도는 번식을 위한 SNP 마커의 식별에 용이하게 사용될 것이며, 더 많은 품종의 염소를 resequencing해야 할 것이다.
Capra hircus 종은 반추동물 중 처음으로 해독된 동물로, 해독 정보를 이용하여 반추동물과 반추동물이 아닌 동물을 기능 유전자로 구분할 수 있게 되었다. 뿐만 아니라 생물 의학 모델 및 생물 반응기로 염소의 유용성을 개선할 수 있게 된 것이다. 더 나은 캐시미어를 생산하기 위해 염소 사육에 대한 마커로 사용될 이번 연구를 토대로 다양한 품종의 de novo sequencing이 필요하다는 것이 입증되었다.
- Contig, Scaffold, super-scaffold : 중복 부위를 갖는 DNA 단편들의 집합.
- 공직선성(colinearity) : 어떤 유전자의 뉴클레오티드 배열이 그것에 의해 결정되는 폴리펩티드 사슬의 아미노산 배열에 대응한다고 하는 개념
- RHMap : radiation hybrid maps. 방사선을 이용한 염색체의 작은 조각contig, scaffold, super-scaffold)의 순서를 정해 재정렬한 지도
- Synteny : 염색체 상의 유전자 구성의 상동성.
- 4DTv(fourfold degenerate site) : 전장유전체 복제 또는 종 간 특징을 각 유사 유전자 간 거리로 측정하는 방법
참고문헌
Sequencing and automated whole-genome optical mapping of the genome of a domestic goat (Capra hircus)
http://www.nature.com/nbt/journal/vaop/ncurrent/full/nbt.2478.html
The Pfam protein families database.
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2238907/
The PRINTS database: a fine-grained protein sequence annotation and analysis resource–its status in 2012.
http://database.oxfordjournals.org/content/2012/bas019.abstract
저자
글 : Park.Hyeonji
편집 : Ahn.Kung
키워드 : De novo sequencing, synteny, Capra hircus, BAC sequencing, NGS(next generation sequencing), CFD(channel-forming device), 등